AI从头设计抗体模型未经专门训练就掌握了可开发性特征?
AI从头设计抗体模型未经专门训练就掌握了可开发性特征?

DrugIntel
发布于 2026-02-04 14:22:50
发布于 2026-02-04 14:22:50
作者:Adil Yusuf
“我不想点名其他AI生物科技公司——你们都很熟悉它们——但我感觉它们总是强调‘我们需要更多数据’、‘数据不足’。这有点像在找借口。其实应该做的是改进算法、优化模型。数据已经足够——只要在算法层面足够创新。”——Demis Hassabis
声明:本文更多是随感而发。若读者感兴趣,未来或可整理成正式论文。
过去一年是蛋白质从头设计的突破之年。学术团队、初创企业,甚至某些科技公司(原因何在?)纷纷开发出新模型与工作流程,成功实现了微型结合蛋白、VHH抗体、单链抗体乃至完整单克隆抗体的从头设计。
令人惊讶的是,取得这些成果的并非单一技术路径,而是多种不同方法,每种方法针对不同靶点均能以个位数或两位数的命中率获得理想结合剂。
已有学者对不同方法进行比较(如本文),而新方法仍层出不穷,例如AbSci公司发布的Origin-1模型。关键在于,这不仅是概念炒作。这些模型真实存在,并将深刻改变药物研发的格局!
在众多新方法中,Chai与Latent Labs最新论文中的发现尤为引人注目。Chai-2和Latent-X2生成的抗体不仅结合性能优异——它们更具备了“药物特性”!表达量高、稳定性强、无聚集倾向,且脱靶结合率低。
这类可开发性特征的优化通常耗时数月,是研发中的难点。许多CRO机构和初创企业正将此类多目标优化作为核心卖点。
值得注意的是,Chai和Latent并非首个实现该突破的团队。2024年末,Nabla已证明其JAM-1模型能生成符合“行业标准”可开发性指标的从头设计VHH抗体,而新版JAM-2的表现甚至可能超越Latent X-2与Chai-2。那么为何这些新模型仍引发广泛关注?
Latent团队做出了大胆声明:
“(我们)设计的分子展现出与已获批抗体疗法相当或更优的可开发性特征,包括表达量、聚集倾向、多反应性、疏水性和热稳定性,且无需优化、过滤或筛选……针对TNFL9靶点的代表性从头设计VHH结合剂同时具备高效靶点结合能力与低免疫原性……这些特性直接由模型涌现,证明了零样本分子设计的治疗可行性。”
他们宣称未使用任何可开发性监督数据集进行训练或筛选,但生成的抗体仍具备这些特性。从现有信息看,Chai-2可能也未经可开发性数据训练——这主要基于其团队规模、人员构成及未提及定制数据集的推测。或许他们使用了少量公共数据集作为评分指标,这点尚不明确。
作为参照,Nabla团队历时多年开展了70余项从头设计抗体项目¹,积累了丰富的数据集用于模型训练与对齐。按常规认知,这种数据壁垒正是理想优势。而Chai和Latent未采用类似策略,却取得了具有竞争力的结果。
那么,可开发性是否已成为从头设计模型的“涌现特性”?可开发性竟是免费获得的吗?
但原因何在?机制如何?这究竟意味着什么?
天下没有免费的午餐,对吗?如果模型能胜任此任务,必然存在某种解释。可能性包括:1)现有评估指标本身存在缺陷;2)可开发性特征已编码在训练数据中/可通过训练数据学习;3)某些未公开的“第三种因素”。
评估指标真实可信吗?
在某些游戏中,作弊是最简单的获胜方式。从头设计抗体领域曾出现通过创造性定义宣称胜利的先例,因此有必要对这些指标的可靠性进行复核。
可开发性如何定义?
学界普遍引用Jain等人2017年的论文(https://pubmed.ncbi.nlm.nih.gov/28096333/),该研究系统评估了当时几乎所有临床阶段抗体(CST)的特性。研究者将抗体统一重构成IgG1格式后进行12项生物物理检测,最终将检测指标归纳为5类(笔者认为至少应分为6类):表达水平(HEK滴度)、热稳定性(Tm)、疏水作用(SGAC100、SMAC、HIC)、长期聚集倾向(AS)、抗原混杂性(ELISA、BVP)以及交叉/自相互作用(PSR、CSI、AC-SINS、CIC)。若需了解这些检测详情,可参阅https://www.blopig.com/blog/2017/07/antibody-developability-experimental-screening-assays/。Jain团队将各类特性(表达与热稳定性除外)的后10%标记为警示信号,最终统计警示数量。
该论文旨在建立启发式标准,而非构建严格分类阈值。尽管存在较多混杂因素,但仍可见清晰趋势:后期阶段CST的平均警示数更少。由于这些检测是在所有CST开发完成后才普及的,我们应该未陷入古德哈特定律(Goodhart's law)的陷阱。
诚然,药物开发有时更接近艺术而非纯粹科学,可参考Lipinski五规则。
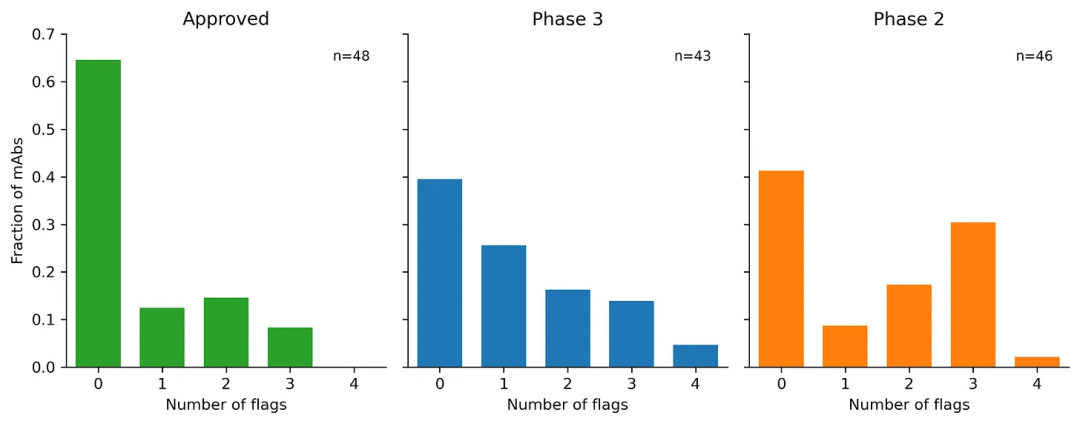
Jain等人2017年的论文
Chai与Latent均尝试用部分CST抗体复现了部分检测,结果令人满意。经尺度调整后,他们各自建立了警示阈值体系。
两家团队涵盖了较全面的可开发性特性。其阈值设定依据虽略显模糊,但整体合理。笔者用参照抗体进行对比后发现,Latent的阈值设定相对宽松,其中Lirilumab的HIC保留阈值尤其难以验证。
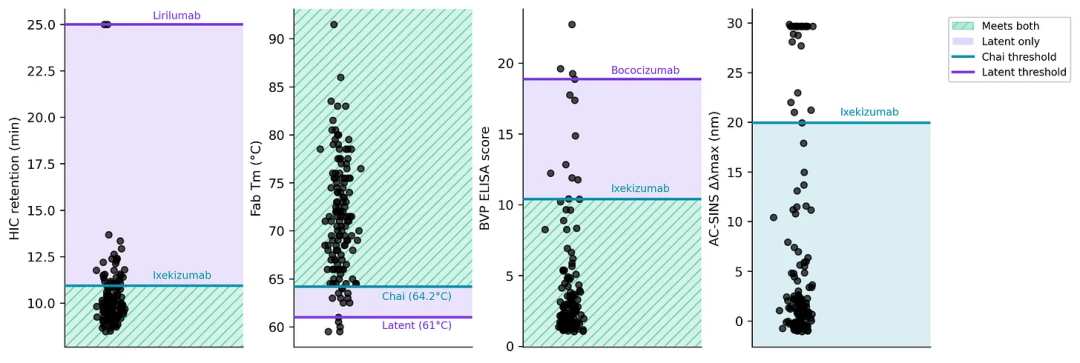
基于参考抗体绘制的Chai-2和Latent X-2可开发性检测阈值与原始Jain 2017数据集的对比图。 Lirilumab的HIC保留时间值设定为25分钟,但在原始研究中该抗体并未被洗脱。Latent X-2未设置AC-SINS的阈值。Tm(热稳定性)分数为精确值,与数据集的映射并非完全吻合。Chai-2和Latent X-2均采用大于或等于90%的单体纯度(通过SEC测定)作为标准,且Latent X-2将此作为额外的警示旗标(flag)阈值。
基于这些阈值,Chai与Latent分别报告其超过85%和80%的设计能在4项指标中获得3-4个“绿色通过”。
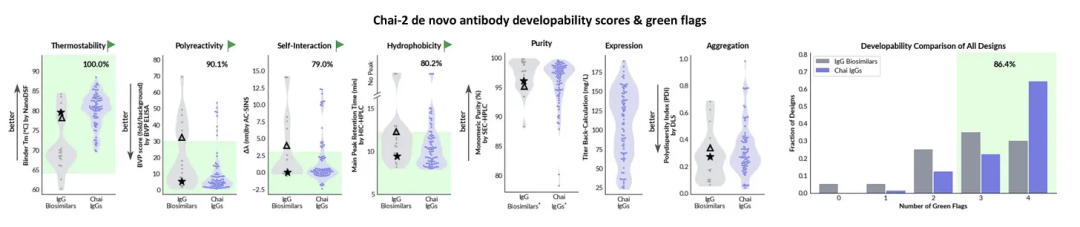
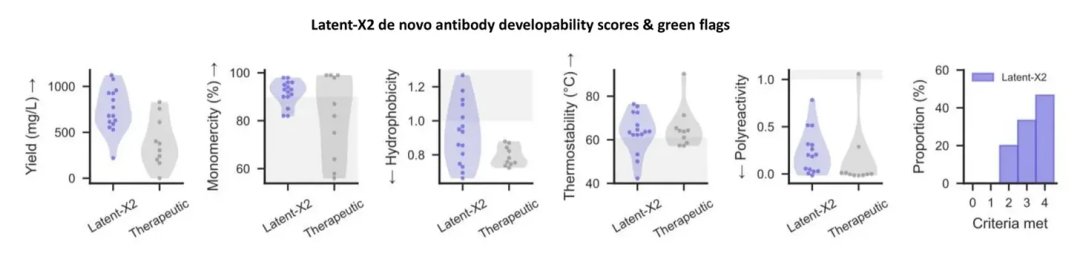
由此可见,检测方法真实有效,阈值设定基本合理,而零样本设计能获得超过80%的通过率确实非常出色。它们是如何实现的?
或许奥秘全在训练数据中?
简而言之:模型只能从训练数据中学习。若它们能进行分布外泛化,则说明这些知识已蕴含在训练数据中。
关于AlphaFold等AI蛋白质模型是否真正学习物理原理的讨论已有很多。这是一个重大论断,需要相应强度的证据支撑。而笔者认为更可能发生的是:训练数据本身包含优质结构。非常优质的结构——其优质程度可能超出人们(至少是我)的认知。
要对蛋白质进行结构表征,首先需要表达该蛋白。表达出的蛋白(及其结合伴侣)必须具备足够稳定性,以承受纯化、冷冻等处理流程。要解析高质量结构,蛋白质还需避免过度聚集。当然,还需要说服资助方投入资源开展实验。因此这些蛋白往往具有某种“重要性”⁴。
这存在显著的选择偏差!
PDB中抗体结构数量有限(约5千至1万个),其中仅少数属于CST/已获批抗体(约200个)。多数结构是否经过可开发性优化尚不明确。仅凭这种选择偏差就足够了吗?
可通过计算评估PDB中所有抗体结构的可开发性来探究。Raybould等人开发的治疗性抗体分析工具2(TAP2)尝试用计算方法复现Jain 2017的核心指标,其警示数量统计显示出相似趋势。笔者在复现TAP2时发现盐桥校正存在一处错误,会影响总警示数。经联系Raybould和OPIG团队,他们正在修正。此处将同时展示修正前后的评分,但总体结论基本不受影响。
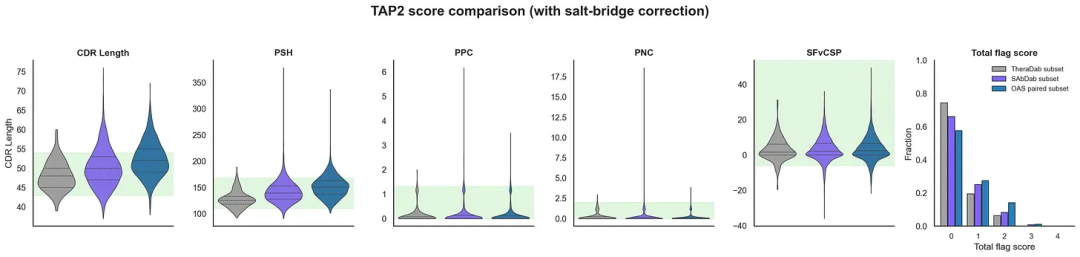
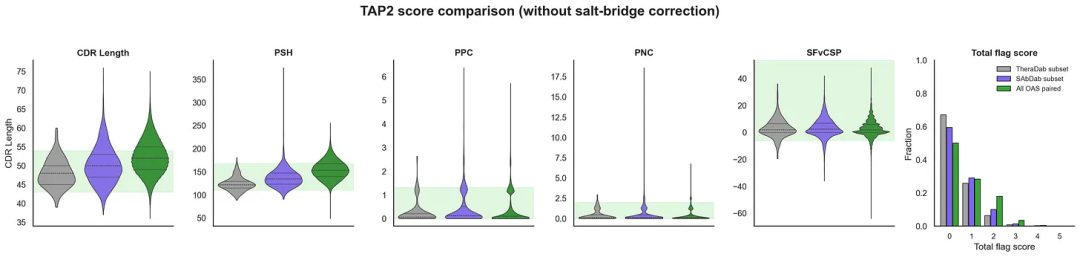
比较治疗性抗体、结构抗体与天然人源抗体的TAP2可开发性评估。 小提琴图展示了五种TAP2指标:CDR长度、表面疏水斑块、正电荷斑块、负电荷斑块以及单链Fv电荷对称参数;绿色阴影区域表示“健康”范围。柱状图展示了总警示旗标分数(每个AMBER/RED旗标计1分)的标准化分布。(上图)包含盐桥校正,使用了来自新冠患者的OAS配对数据集子集(OAS paired subset,蓝色)。(下图)不包含盐桥校正,使用NaturalAb TAP2数据作为天然抗体参照(所有OAS配对序列,绿色)。
基于TAP评分,PDB中的抗体结构(即SAbDab数据集)实际上展现出良好的可开发性,甚至优于OAS配对抗体的天然序列(具有统计学显著性)。即使排除PDB中的CST抗体结构,分别与OAS初始B细胞和记忆/浆细胞来源序列对比,SAbDab仍存在这种偏差(图示略)。这种差异主要源于CDR长度和PSH指标,二者存在相关性。值得注意的是,OAS配对序列本身已具有不错的可开发性评分。虽非严格分析,但这一现象值得关注!
在免疫原性方面,Chai未进行实验评估,而是通过BioPhi/promb工具包计算抗体序列的“人源化程度”,其从头设计序列表现良好。
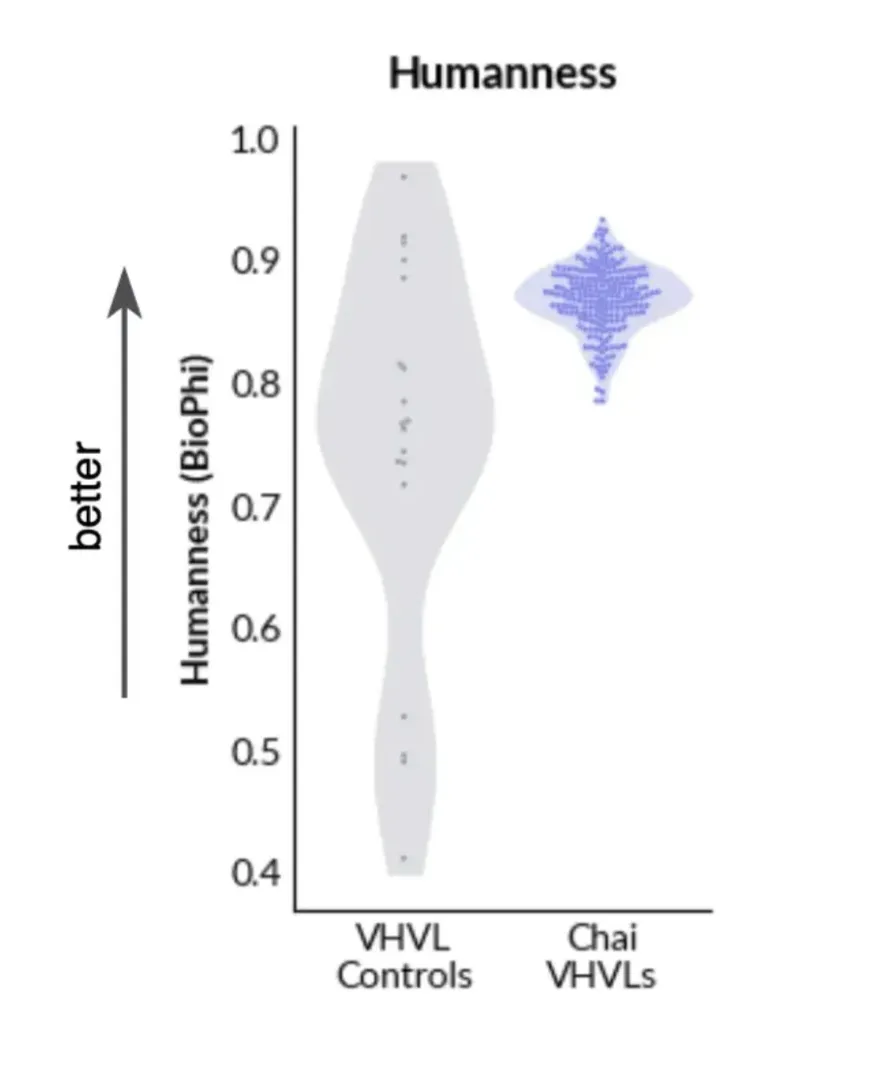
笔者对PDB数据进行了类似分析。如预期所示,OAS序列和CST人源抗体显示高度人源化特征。而PDB中抗体结构的平均人源化程度与CST人源化抗体相当。

各数据库抗体序列的BioPhi/OASis人源化程度评分。 TheraDab:治疗性抗体(按来源分层的子集:人源、人源化、嵌合、鼠源)。OAS:来自Observed Antibody Space的序列(初始B细胞和浆细胞/记忆B细胞子集)。SAbDab:已解析结构的抗体(包含及不包含与TheraDab重叠的部分)。黑色横线表示中位数。
至少在这些计算指标上,PDB中的抗体确实呈现“类药物”特性!
Latent团队则通过实验检测了部分从头设计VHH在10名健康供体原代免疫细胞中的免疫原性。采用T细胞增殖和细胞因子释放检测,结果显示所有VHH在所有供体中均未引发异常反应!或许Chai的抗体也将具有低免疫原性……
PDB抗体结构对良好可开发性特征的偏好,能否解释这些模型的涌现特性?也许OAS的合成结构本身已足够优质?
存在未公开的“第三种因素”?
我们尚不清楚具体实现细节。或许存在某些未公开的“秘密技术”提升了抗体的可开发性。
仅使用优质框架序列似乎难以产生如此效果,因为部分可开发性特性与框架无关(如聚集性、多反应性)。Chai指出其采用VH3-23和VH3-66框架——Jain 2017研究显示这些框架具有良好的生物物理特性。Latent则在生成从头设计抗体时使用了5个已验证框架序列。若仅凭框架选择就能实现目标,业界早该普遍采用这些优质框架,而无需大量优化工作。何况Chai-2抗体至少在热稳定性上超越了框架匹配的对照样本。虽然存在更细致的反驳观点,但笔者尚未找到令人信服的论据。
Nabla宣称通过70余项从头设计抗体项目训练JAM模型。Chai或Latent是否秘密获取了大规模可开发性数据集?能否用小型可开发性数据集和计算指标有效对齐从头设计模型或评估抗体?是否在合成数据生成或新型训练/推理技术上有所突破?还是存在其他因素?
乐观来看,若无需特殊数据,开源模型的前景将更加光明。或许Boltzgen抗体(或其他开源模型)已具备良好的可开发性特性!
结论
在现有合理的可开发性指标/阈值下,Chai和Latent生成的从头设计抗体均展现出“良好”的可开发性。它们似乎未使用(或仅使用极少量)可开发性数据进行训练、过滤或筛选。
可能原因包括:1)结构数据中存在的可开发性偏差在模型扩展过程中被学习;2)存在未公开的核心技术。
据笔者推测,以下因素的综合作用将我们推入了优质区间:精心选择优势表位、训练数据偏差、结合力评分、风险特征过滤(及可开发性?)、优质框架序列的运用。
或许Demis的观点是正确的。我们可能不需要更多数据,而是需要更优的算法与模型。药代动力学/药效学特性能否从现有结构数据中学习?还有哪些潜力尚未挖掘?Isomorphic团队的研究方向引人遐想……
抗体发现与优化全面GPU化的时代正在临近。我们仿佛跨越了某个阶段直接抵达此处,这值得深入思考。虽无法确定具体机制,但若可开发性真能成为 免费赠品 ,无疑将是重大利好。
参考链接:https://ayusuf.substack.com/p/developability
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2026-01-30,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

