内容复习--分子niche与细胞niche的区别
原创内容复习--分子niche与细胞niche的区别
原创
追风少年i
发布于 2026-01-07 11:04:53
发布于 2026-01-07 11:04:53
作者,Evil Genius
先给大家一张分析的思路图
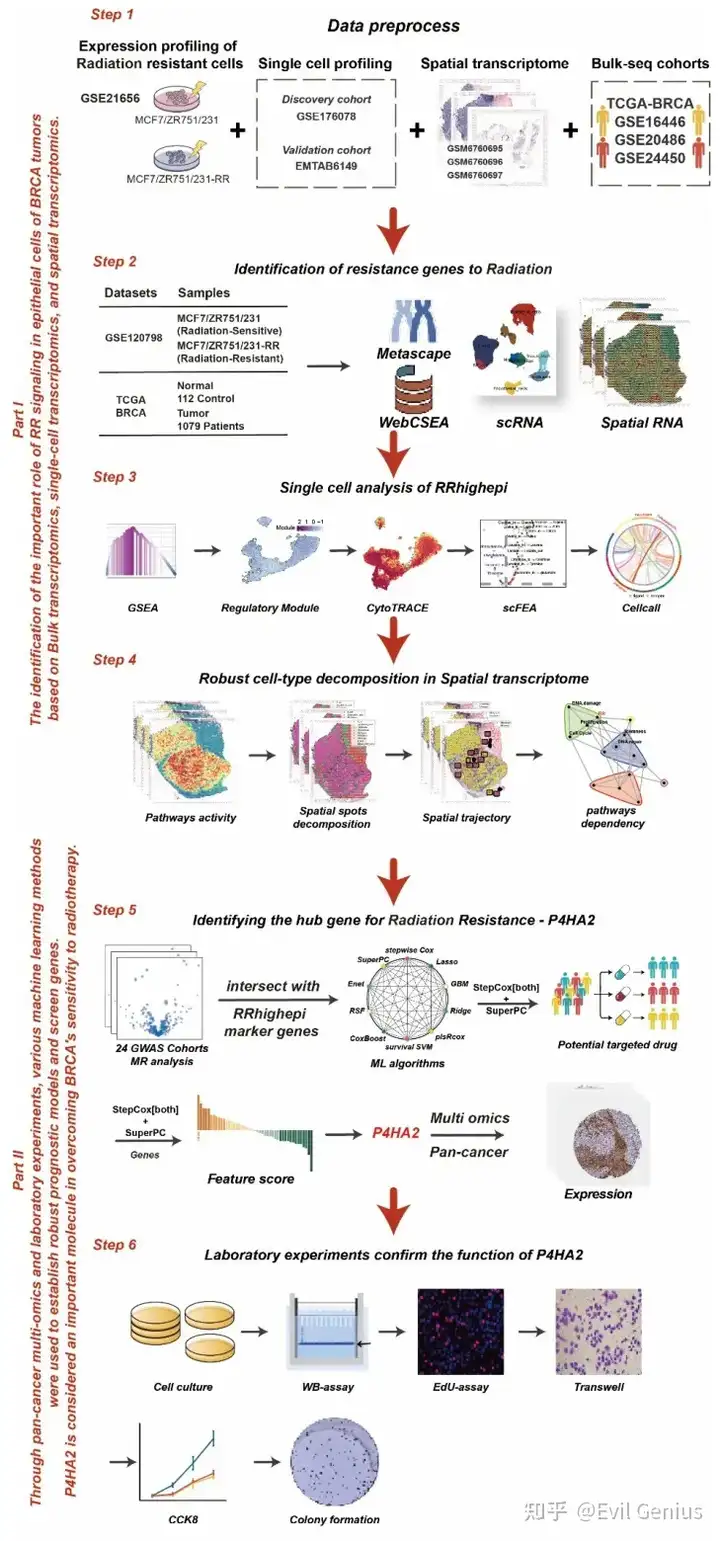
其实很多学员问,说文章对visium的注释直接联合即可,后续分析细胞niche才会多样本比较, 而且用的是细胞矩阵,那么多样本的基因矩阵整合还有没有必要做呢?
其实问题的核心在于分子niche与细胞niche的差异点在哪里以及背后的生物学意义。
我们来简单判读一下
首先是细胞niche
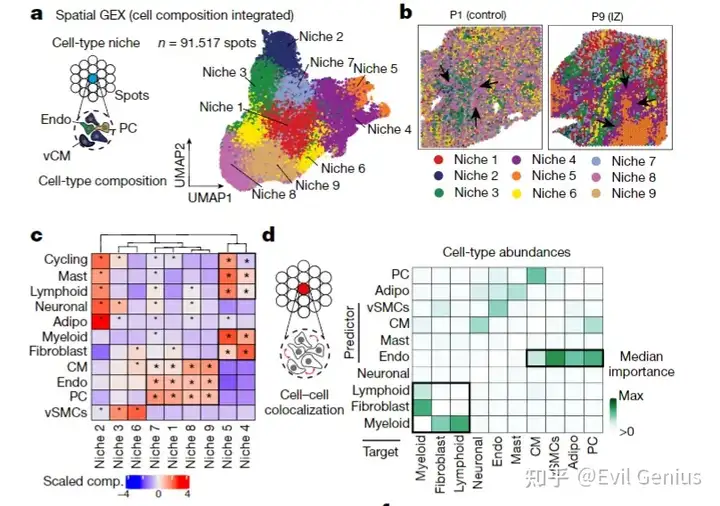
首先看分析内容
对所有样本的所有空间 spots,基于其细胞类型组成向量(即每种细胞在该 spot 的丰度)进行无监督聚类;
聚类得到 cluster,每个cluster代表一种重复出现的细胞组合模式,被定义为一个 “细胞类型微环境”(cell-type niche)。
通过富集分析,将每个 niche 与特定功能或病理状态关联(如肌源性、炎症性、纤维化等)。
结合空间位置可视化,确认 niche 在组织中的空间分布特征(如均匀分布 vs 局部富集)。
进一步分析依赖关系
使用 MISTy 模型 分析:一个 spot 中某细胞类型的丰度是否可由其邻域内其他细胞的组成预测;
评估不同空间尺度(自身、局部邻域、扩展邻域)下的细胞间依赖性。
其核心生物学意义:
1、揭示组织的功能结构单元
这些 niche 被视为心肌组织中共享的、可重复的结构-功能模块(“building blocks”),在不同个体和切片间保守存在,便于跨样本比较。
2、关联病理状态
特定 niche 与疾病状态高度相关:
Niche 5(髓系+淋巴细胞富集)→ 炎症微环境,在缺血区域特异性富集;
Niche 4(成纤维细胞为主,伴髓系细胞)→ 纤维化微环境,反映心肌修复/瘢痕形成过程。
3、揭示细胞间互作与依赖关系
内皮细胞是多种血管相关细胞(vSMCs、周细胞、心肌细胞)丰度的关键预测因子 → 支持血管网络在心肌组织架构中的核心作用;
髓系细胞与成纤维细胞相互依赖 → 反映免疫-基质交互在纤维化中的机制(如巨噬细胞激活成纤维细胞)。
4、整合稀有细胞类型的空间生态位
成功识别包含血管平滑肌细胞(vSMCs)、脂肪细胞、增殖细胞等稀有群体的 niche,说明该方法能捕捉低丰度但功能重要的细胞共定位事件。
5、为分子机制研究提供空间上下文
建立了“细胞共定位图谱”,使得后续可在特定 niche 内进行差异表达、通路富集或配体-受体互作分析,实现从空间结构到分子机制的桥梁。
接下来看分子niche
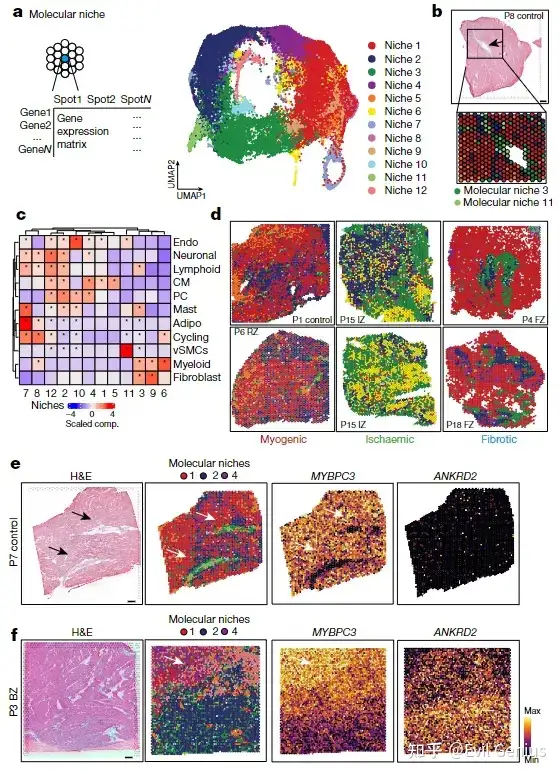
分子niche是基于空间 spots 的基因表达谱进行聚类,定义了一组 “分子微环境”(molecular niches)。
核心意义在于
1. 揭示细胞功能状态的异质性,超越细胞类型组成
即使在相同细胞类型(如心肌细胞)占主导的区域,基因表达谱的差异可定义出多个分子 niche;
这些 niche 反映了细胞在代谢、电生理、结构和应激响应等方面的功能亚态(functional states),而非仅仅是“谁在那里”。
2. 捕捉组织重构中的细微病理变化
分子 niche 能区分细胞组成相似但功能状态不同的区域(如对照 vs 远端区 vs 边缘区),而传统基于细胞类型的 niche 无法做到;
揭示了看似“正常”的远端心肌其实已发生分子层面的适应性改变。
3. 关联空间位置与疾病进程
特定分子 niche 与特定病理区域高度共定位:
炎症/纤维化相关 niche → 缺血或瘢痕区域;
应激型心肌细胞 niche → 梗死边缘区(损伤前沿);
稳态型心肌细胞 niche → 对照或远端区。
从而将空间位置、组织结构与分子功能三者统一。
总结一下就是:分子 niche 分析通过解码空间 spots 的基因表达程序,揭示了心肌组织在梗死后不仅“细胞种类变了”,更关键的是“同一种细胞的功能状态也发生了精细重编程”,从而在分子层面刻画了心脏损伤、适应与重构的动态图谱。
综合比较一下
比较维度 | 细胞 Niche(Cell-type Niche) | 分子 Niche(Molecular Niche) |
|---|---|---|
定义基础 | 基于每个空间 spot 中各类细胞的相对丰度或组成(如心肌细胞、成纤维细胞、免疫细胞等的比例) | 基于每个空间 spot 的全基因表达谱(不依赖细胞类型注释,直接聚类基因表达模式) |
数据输入 | 反卷积后的细胞类型丰度矩阵(如 cell2location 输出) | 空间转录组原始或标准化后的基因表达矩阵 |
核心目标 | 描述“哪些细胞共存于同一微环境”,识别重复出现的细胞组合模式 | 描述“哪些分子程序在局部区域共同激活”,识别具有相似转录状态的功能区域 |
分辨率层次 | 细胞组成层面(“谁在那里”) | 功能状态层面(“它们在做什么”) |
能否区分相同细胞类型的不同状态? | ❌ 不能 —— 若两个区域都富含心肌细胞,即使其功能状态不同,也会被归为同一细胞 niche | ✅ 能 —— 可将心肌细胞主导但分子特征不同的区域(如稳态 vs 应激)划分为不同分子 niche |
典型应用场景 | - 识别炎症、纤维化、血管等宏观组织结构- 跨样本比较组织构成- 构建细胞互作空间图谱 | - 发现细胞功能异质性- 捕捉早期/细微病理变化- 关联通路活性与空间位置 |
生物学意义 | 揭示组织的结构性单元(如“免疫-成纤维细胞微环境”),反映细胞共定位与潜在互作 | 揭示组织的功能性单元(如“钙调控心肌微环境”),反映局部分子程序与细胞状态重编程 |
局限性 | 无法捕捉同类型细胞的功能差异;依赖反卷积质量与细胞注释准确性 | 难以直接解释驱动因素(需后续注释);可能混杂多种细胞类型的信号 |
互补性 | 回答“组织由哪些细胞模块构成?” | 回答“这些模块内部在执行什么分子功能?” |
所以综合分析才能得到全面、合理的结果。
生活很好,有你更好
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
评论
登录后参与评论
推荐阅读
目录

