基因组+单细胞--弥漫性大B细胞淋巴瘤的生物学变异轴向
原创基因组+单细胞--弥漫性大B细胞淋巴瘤的生物学变异轴向
原创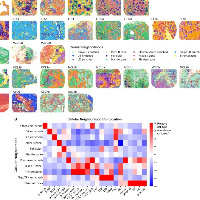
追风少年i
发布于 2026-01-19 10:05:27
发布于 2026-01-19 10:05:27
作者,Evil Genius
分享文章之前,大家对基因组 + 单细胞的联合分析思路有了解了么?
单细胞分析的CNV可以和WES的CNV分析相互对应么?
比较维度 | WES-CNV (基于Bulk WES) | scCNV (基于scRNA-seq,如inferCNV分析) | 互补与验证关系 |
|---|---|---|---|
检测本质 | 直接检测:对肿瘤组织混合DNA进行测序,通过计算测序深度变异直接得出拷贝数信息。 | 间接推断:利用scRNA-seq数据中基因的表达量偏差(与“正常参照细胞”相比)来推断DNA拷贝数的变化。前提是CNV会导致基因表达量的系统性偏移。 | 根本性差异:WES看的是DNA本身的数量,是原因;scRNA-seq推断看的是基因表达水平的间接表现,是结果之一。两者并非检测同一物质。 |
分辨率 | 基因组分辨率高:可精确定位到具体的外显子/基因区域(通常几十kb到Mb级)。细胞分辨率低:结果为所有细胞CNV状态的加权平均。 | 基因组分辨率低:通常只能识别较大的染色体区段(整臂或整条染色体水平),分辨能力有限。细胞分辨率极高:可展示细胞群体水平的CNV模式差异,识别恶性细胞亚群。 | 信息互补:WES是“高精度地图”,精确定位断点和拷贝数。scRNA-seq推断是“细胞分布热力图”,显示哪些细胞群体具有相似的大规模拷贝数变异模式。 |
主要优势 | 1. 金标准与定量准:是临床和科研中鉴定CNV的可靠方法,可估计绝对拷贝数。2. 灵敏度高:能发现低频、小片段CNV。3. 技术成熟:流程标准化,可重复性好。 | 1. 解锁肿瘤异质性:核心价值在于无需预先分离,即可将恶性细胞(显示大规模CNV)与正常微环境细胞(无大规模CNV)区分开,并进一步划分恶性亚克隆。2. 关联表型:CNV推断与同一细胞的全转录组数据天然整合,可直接分析CNV对下游基因表达网络的影响。 | 优势互补:WES提供准确的变异列表;scRNA-seq推断揭示这些变异在细胞生态系统中的分布和功能关联。 |
主要局限 | 1. 无法解析异质性:无法区分一个CNV是存在于所有肿瘤细胞,还是仅存在于一个亚克隆。2. 受肿瘤纯度影响大。 | 1. 间接推断,噪音大:受基因表达生物学波动、技术噪音影响大,假阳/阴性较高,不能作为绝对定量的依据。2. 仅适用于大规模CNV:对局灶性、小片段CNV不敏感。3.依赖参考细胞:需要从同一数据中定义“正常二倍体”细胞作为参照,若参考细胞选择不当,结果会失真。 | 相互验证与校正:1. WES验证scRNA-seq推断:用WES确认的、明确的大片段CNV(如chr8q扩增、chr13q缺失)作为“地标”,检查inferCNV结果中是否在同一位置出现一致信号。这评估了推断的可靠性。2. scRNA-seq推断解析WES结果:当WES检测到中等强度CNV信号时,inferCNV可揭示这是“广泛弱改变”还是“局部强克隆”,从而解释WES信号的细胞来源。3. 联合分析提升价值:将WES鉴定的关键驱动CNV(如MYC位点)作为重点,在inferCNV图谱中观察其在不同亚克隆中的表达影响,实现“基因型-细胞表型”的整合。 |
今天我们就来分享一篇这样的文章
知识积累
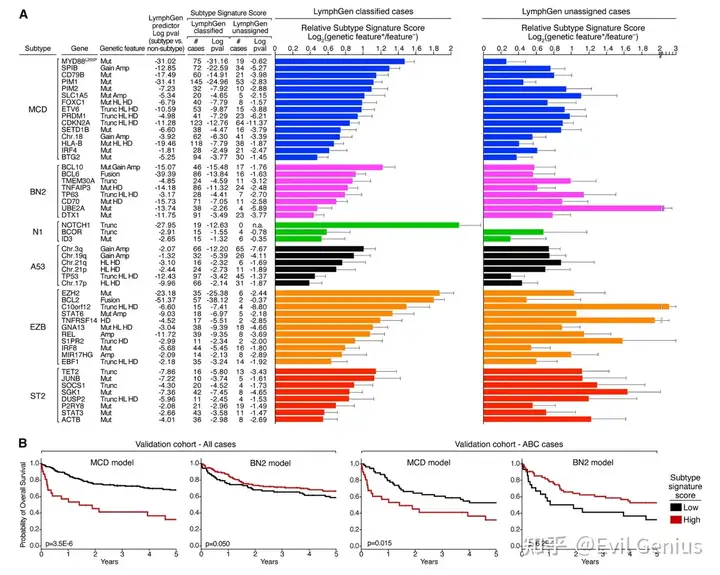
弥漫性大B细胞淋巴瘤(DLBCL)对化疗和靶向治疗的反应差异与其基因型和表型的异质性密切相关。研究最初基于细胞起源(COO)将其分为两类:GCB亚型(基因表达类似正常生发中心B细胞)和ABC亚型(类似B细胞受体激活的外周血B细胞)。在此基础上,遗传谱分析进一步定义了7种DLBCL遗传亚型(可通过LymphGen算法划分),约62%病例可归类为MCD、BN2、N1、A53、EZB或ST2亚型,其中EZB又细分为EZB-DH(伴BCL2和MYC易位)与EZB-nonDH。
遗传亚型与COO分类高度相关:EZB最具GCB特征,MCD最接近ABC表型。此外,N1和A53多呈ABC表型,ST2多为GCB表型,而BN2表型多变且常被COO分析归为“未分类”。
遗传亚型对临床具有重要价值:
预后分层:可区分R-CHOP化疗后不同风险的患者群体;
指导靶向治疗:
MCD型对BTK抑制剂伊布替尼敏感,且联合R-CHOP可显著改善MCD/N1型患者生存;
多靶点联合方案ViPOR在MCD和EZB-DH型中疗效尤为突出。
为深入解析DLBCL生物学特性,研究通过单细胞图谱聚焦三个核心问题:
(1)遗传亚型是否具有独特的恶性细胞基因特征和肿瘤微环境?
(2)DLBCL是单克隆还是存在遗传/表型异质的亚克隆?
(3)各亚型恶性细胞的特征性转录因子网络如何与正常B细胞分化调控相关联?
DLBCL的分子分型逐步精细化,从COO到遗传亚型的演变不仅揭示了疾病异质性,更为预后评估和靶向治疗选择提供了关键依据。单细胞研究将进一步解析亚型特异的生物学机制及克隆多样性。
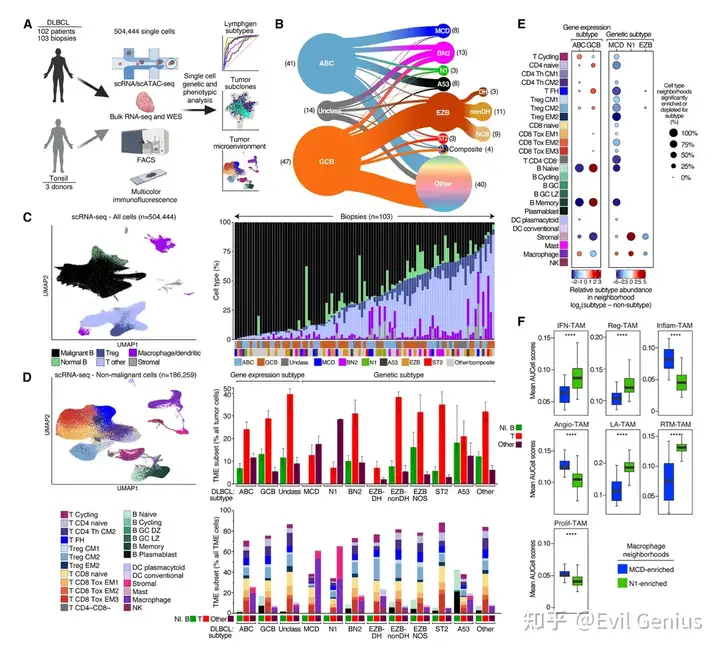
结果1、单细胞RNA测序、单细胞ATAC测序及大块肿瘤全外显子组与RNA测序
恶性细胞识别与验证
通过单细胞RNA数据推断拷贝数变异,成功区分恶性B细胞与正常B细胞。
推断的CNV与大块肿瘤WES结果一致,证明单细胞推断的可靠性。
利用克隆性免疫球蛋白序列进一步确认恶性细胞身份。
肿瘤微环境细胞图谱
鉴定了正常B细胞(6个亚群)、T细胞(12个亚群)以及先天免疫与间质细胞(6类)。
EZB-DH亚型的TME最贫乏,T细胞数量最少;而EZB-nonDH亚型具有最丰富的T细胞浸润,提示二者不同的病理生物学特征。
MCD亚型的CD8⁺初始T细胞耗竭特征最显著,且巨噬细胞富集(尤其是促血管生成亚群Angio-Mac)。
N1亚型富集间质细胞和两类巨噬细胞:干扰素激活型(IFN-Mac)与脂质相关型(LA-Mac)。
微环境与遗传亚型的统计学关联
通过空间邻域分析发现:
EZB-nonDH富集滤泡辅助性T细胞和调节性T记忆细胞。
MCD和N1的巨噬细胞虽同属一大类,但具有不同的功能亚群特征。
结果2、弥漫性大B细胞淋巴瘤遗传亚型的表型特征
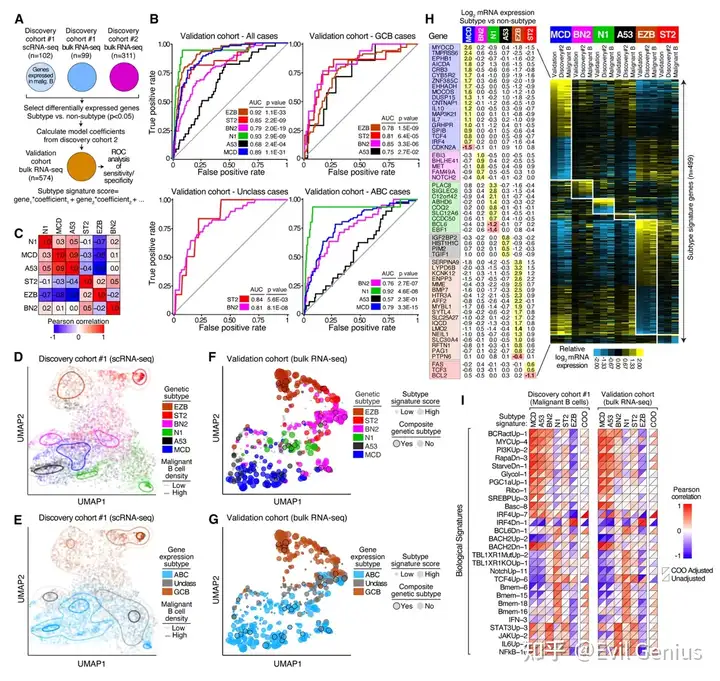
恶性细胞的表达谱具有亚型特异性:基于单细胞亚型评分进行的聚类分析显示,不同遗传亚型的恶性细胞形成了相对独立但部分重叠的集群。
模型整合了细胞起源信息:基于单细胞数据的COO分型(ABC/GCB/未分类)结果与遗传亚型评分聚类结果高度一致,表明所建模型包含了细胞起源信息。
模型揭示未分型病例的潜在归属:即使在LymphGen算法中未能归入明确亚型的“其他”病例,在UMAP图谱上也倾向于与某个遗传亚型重叠,提示其具有相似的生物学背景。
复合亚型病例的生物学倾向:被LymphGen判断为具有多种亚型可能性的复合DLBCL病例,其表达谱通常与其遗传主导亚型(概率最高者)的病例聚集在一起。
结果3、通过基因表达特征揭示的DLBCL遗传亚型生物学
一、亚型特征基因与关键通路
MCD/A53亚型:
表达谱富集于ABC/活化B细胞特征,涉及慢性活跃BCR信号(BCRactUp-1)、MYC活性、PI3K-mTOR通路、糖酵解与核糖体生物合成。
关键转录因子IRF4/SPIB/TCF4活性高,驱动其靶基因(如AICDA、IL-10)表达,并倾向浆细胞分化轨迹(与浆母细胞特征Basc-8正相关)。
MCD因CDKN2A频繁纯合缺失而表达低。
BN2亚型:
特征为NOTCH2突变/高表达,驱动NOTCH靶基因(如EBI3、NOTCH2)表达。
同样显示慢性活跃BCR信号。
N1亚型:
特征为NOTCH1突变,驱动相应靶基因表达。
表达谱与记忆B细胞特征强相关,且与浆细胞轨迹负相关。
BCL6表达下调,BACH2与TCF4活性增强,符合其记忆B细胞表型。
EZB亚型:
表达正常生发中心B细胞标志物(如CD10、LMO2)。
BCL6表达上调,IRF4活性低,抑制浆细胞分化。
PTPN6(SHP-1)表达低,有利于持续BCR信号。
ST2亚型:
多为GCB表型但缺乏部分GC标记(如CD10)。
NF-κB与JAK-STAT3信号活性高(与IκBα、SOCS1失活一致),BCL2表达低,可能解释其对化疗的敏感性。
特征的可重复性与应用价值
高度可重复:89%的特征基因在独立验证队列中重现。
解析未分型病例:即使LymphGen未明确分型的病例,其表达谱也常与某一特定亚型重叠,提示特征模型能揭示其生物学归属。
关联遗传异常:具有特定遗传异常(如NOTCH2突变)的病例,其对应亚型特征评分显著更高,且该规律在已分型和未分型病例中均成立。
预后关联
特征评分与已知生存关系一致:ABC相关亚型(MCD, BN2, N1, A53)特征与不良预后相关,而GCB相关亚型(EZB, ST2)特征与良好预后相关。
在ABC病例中,高MCD评分提示不良预后,高BN2评分则预示较好预后。
各DLBCL遗传亚型具有独特的、可重复的基因表达程序,这些程序由特定的驱动突变/通路(如NOTCH、BCR、IRF4)和转录因子网络塑造,并决定了细胞状态(如活化B细胞、记忆B细胞、生发中心B细胞)与临床预后。
结果4、DLBCL肿瘤中表型不同的遗传亚克隆
普遍存在:通过推断的拷贝数变异(CNV)模式,在大多数肿瘤(79%) 中检测到多个(2-5个)遗传亚克隆,且与DLBCL亚型或既往治疗无关。
可靠性验证:大亚克隆的CNV谱在bulk肿瘤测序数据中得到证实,支持单细胞推断的可靠性。
亚克隆表型差异的量化——六大功能模块
通过计算亚克隆与肿瘤内所有恶性细胞平均表达的“偏差评分”,将差异表达特征聚类为六个功能模块:
B细胞分化相关(四个):
生发中心B细胞功能模块:富集GC B细胞标志(如CD10)及DLBCL抑癌基因。
记忆B细胞功能模块:富集记忆B细胞标志、趋化因子受体(CCR6, S1PR1)、静息调节因子(KLF2)及抑制性受体(SIGLEC6)。
浆细胞功能模块:包含浆细胞关键调控因子(PRDM1、IRF4与XBP1靶基因)。
泛B细胞功能模块:包含B细胞各阶段共表达基因(如PAX5, CD20, CD22)。
恶性增殖/代谢相关(两个):
细胞周期功能模块:增殖细胞高表达。
细胞生长功能模块:调控糖酵解、有氧代谢、蛋白质周转等支持细胞增大的基因;显著富集MYC靶基因(24%),包括多个直接靶点。
三、功能模块的生物学意义
B细胞分化功能模块在正常扁桃体B细胞亚群中特异地区分不同分化阶段。
细胞周期与细胞生长功能模块在正常增殖最活跃的GC暗区细胞中表达最高。
细胞周期与细胞生长功能模块虽相关,但表达模式可区分。
四、肿瘤内亚克隆的表型轨迹
功能模块差异:同一肿瘤内的不同亚克隆在六大功能模块的表达上呈现显著差异。
多样化的克隆轨迹:
常见轨迹:亚克隆保持相同的B细胞分化功能模块,但细胞周期/生长主题表达量不同(反映增殖/代谢异质性)。
其他轨迹:亚克隆间呈现浆细胞 vs. 记忆B细胞,或生发中心B细胞 vs. 记忆B细胞等功能模块的转换。
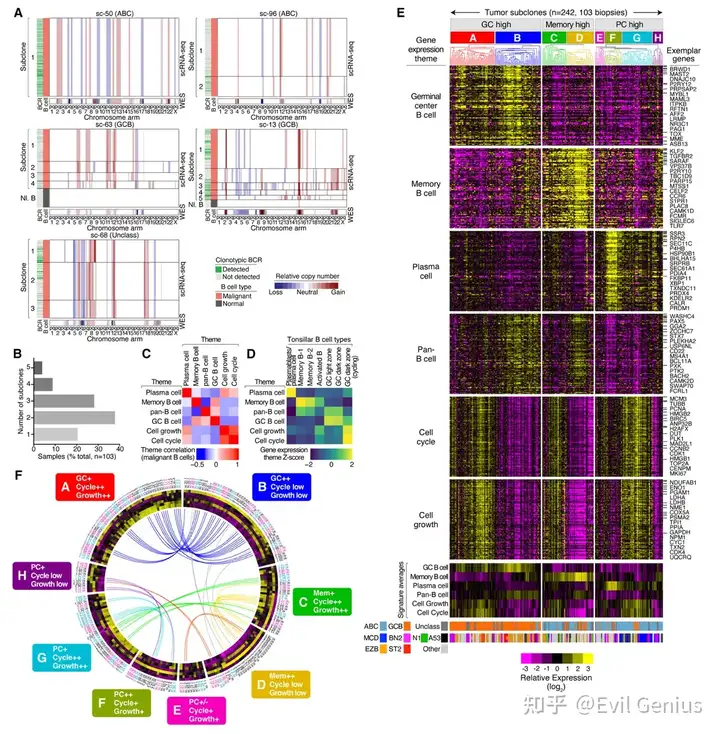
特征模块定义主要表型变异:
每个特征模块的表达都特异性地富集于特定的UMAP细胞cluster,表明这些模块成功捕捉了DLBCL恶性细胞表型变异的主要维度。
相比之下,遗传亚型在恶性细胞中的分布更为均匀,与UMAP簇的对应关系不明显。这说明遗传亚型特征与细胞表型特征模块分别刻画了恶性细胞异质性的不同层面。
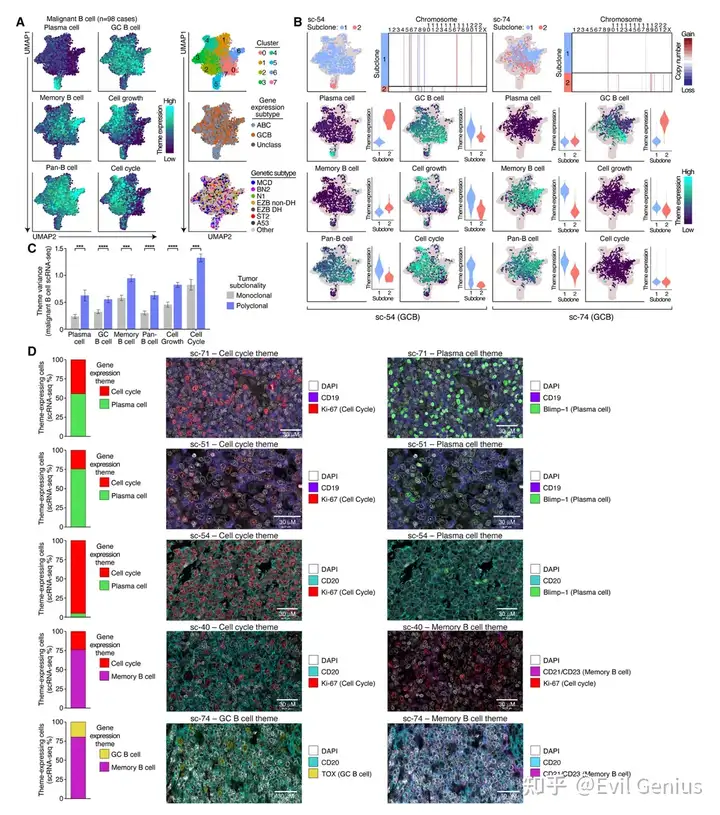
特征模块揭示肿瘤内亚克隆结构:
通过可视化具体肿瘤(如sc-54, sc-74)中不同亚克隆的细胞在UMAP上的位置,证实了亚克隆间存在明显的表型分离。
示例:
GCB肿瘤sc-54:亚克隆1高表达生发中心B细胞、细胞周期和细胞生长模块,富集于UMAP cluster 0和cluster 3;而亚克隆2高表达浆细胞模块,富集于UMAP cluster 5。
GCB肿瘤sc-74:亚克隆1表达记忆B细胞模块(富集于cluster 6),亚克隆2表达生发中心B细胞模块(富集于cluster 0)。
亚克隆结构贡献主要表型变异:
与单克隆肿瘤相比,存在亚克隆的肿瘤在各个特征模块的表达上表现出显著更大的异质性。
即使在单克隆肿瘤内,特征模块表达也存在差异,但程度较轻,其中细胞周期和记忆B细胞模块的变异度最大。
多重免疫荧光成像验证:
使用针对特征模块标志蛋白的抗体进行多色免疫荧光成像,在组织原位验证了预测的肿瘤亚克隆。
表型-蛋白对应验证:
浆细胞 vs. 增殖细胞:在sc-51等肿瘤中,成像显示存在高表达浆细胞标志Blimp-1(伴CD19表达降低、核偏位)的细胞亚群,与高表达细胞周期标志Ki-67的增殖亚群互不重叠,符合Blimp-1抑制增殖的生物学特性。
记忆B细胞 vs. 增殖细胞 / 生发中心B细胞:在sc-40和sc-74肿瘤中,成像分别证实了记忆B细胞标志(CD21, CD23)与Ki-67、或与生发中心B细胞标志(TOX)存在于不同的空间亚群中。
结果5、基因表达模块与淋巴瘤发病机制和生存的关联
模块表达的亚型偏好与临床预后
亚型偏好性:
浆细胞模块:在BN2和ST2亚型中上调。
记忆B细胞模块:在N1亚型中显著高表达。
生发中心B细胞模块:在EZB(包括DH和非DH)亚型中更高。
细胞周期与细胞生长模块:在MCD和EZB-DH中上调,在N1和EZB-nonDH中下调。
模块与遗传亚型的关系:
方差分析表明,特征模块无法完全解释遗传亚型特征的全部变异(未解释残差:EZB 17% 至 BN2 79%),证明遗传亚型特征包含了模块之外的独立生物学信息。
预后关联:
生发中心B细胞模块与良好总生存期相关。
细胞生长模块与不良生存期显著相关,而细胞周期模块无显著预后意义。这凸显了两者在生物学上的差异,尽管表达中度相关。
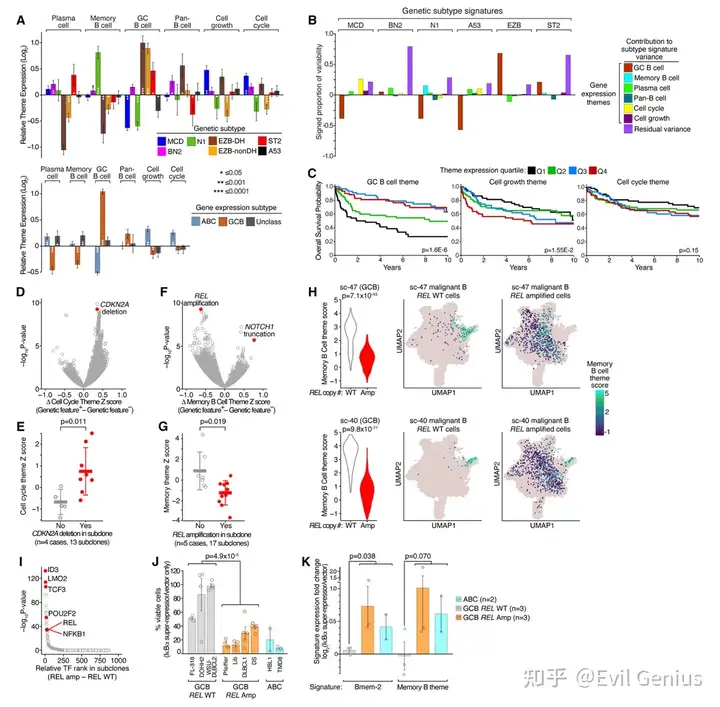
二、驱动模块表达的遗传改变
CDKN2A缺失 → 细胞周期模块高表达:
CDKN2A(编码p16抑癌蛋白)的缺失与细胞周期模块的高表达显著相关,在单细胞亚克隆水平得到验证。
NOTCH1截短突变 → 记忆B细胞模块高表达:
这与NOTCH1突变定义N1亚型的发现一致。
REL扩增 → 记忆B细胞模块低表达(关键新发现):
意外发现:REL基因(编码NF-κB亚基c-Rel)的扩增与记忆B细胞模块的低表达显著相关。
单细胞验证:携带REL扩增的亚克隆,其记忆B细胞模块的表达显著低于无扩增的亚克隆。
表观遗传机制:scATAC-seq分析显示,REL扩增的亚克隆中,开放染色质区域显著富集NF-κB结合基序。
三、REL扩增的功能验证:阻断终末分化
生物学假说:REL扩增在GCB肿瘤中常见(20-25%),可能通过阻断生发中心B细胞向记忆B细胞的终末分化来促进淋巴瘤发生。
功能实验:
在REL扩增的GCB细胞系和具有核REL活性的ABC细胞系中,通过表达不可磷酸化的IκBα超级抑制因子来抑制NF-κB活性,会导致强烈的细胞毒性。
该操作在同样细胞系中诱导了记忆B细胞特征基因和记忆B细胞模块的表达。
在REL野生型的GCB细胞系中,上述效应均不显著。
结果6、正常B细胞分化过程中的基因调控网络
SCENIC+算法。
eRegulon在B细胞分化关键阶段的特异性活性
分析揭示了不同eRegulon在B细胞分化关键阶段具有特异性活性:
生发中心区室:
TCF3 eRegulon在暗区活性更高,符合其促进暗区B细胞增殖的作用。
BACH2 抑制性eRegulon在GC B细胞中低表达,与其作为促进GC功能的转录抑制因子角色一致。
浆细胞:
显著富集 IRF4、XBP1、PRDM1 的激活型eRegulon,分别对应其驱动浆细胞分化、建立分泌表型和双向调控基因的功能。
记忆B细胞:
富集 KLF2 和 STAT1 的激活型eRegulon,与其维持B细胞静息状态和造血干细胞功能的作用相符。
eRegulon动态变化描绘分化轨迹
通过拟时序分析,描绘了三条主要分化路径中eRegulon的梯度变化:
路径#1(向增殖性中心母细胞分化):静息B细胞 → 中心细胞 → 中心母细胞。伴随 TCF3 和 MYBL1 eRegulon活性的变化。
路径#2(向浆细胞分化):静息B细胞 → 中心细胞 → 浆细胞。伴随 XBP1 和 PRDM1 eRegulon活性的梯度上升。
路径#3(向记忆B细胞终末分化):初始B细胞 → 中心细胞 → 记忆B细胞。 KLF2 和 ETV6 eRegulon活性在初始细胞中高,在中心细胞中下降,在记忆B细胞中再次升高。
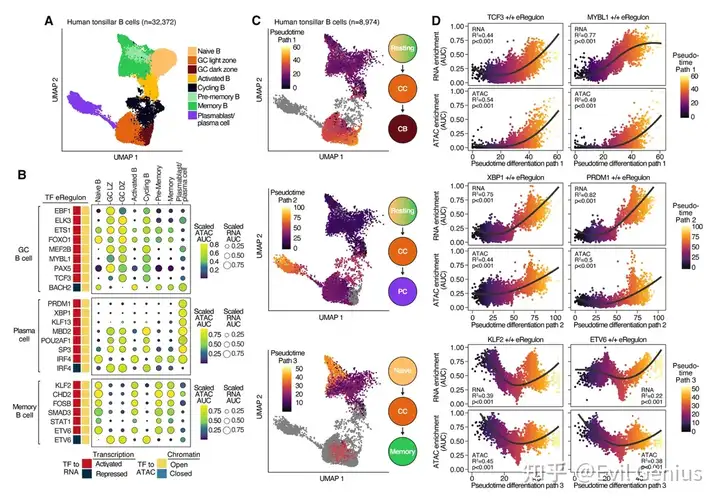
结果7、与DLBCL生物学主题和遗传亚型相关的表观遗传图谱
通过整合ATAC-seq与转录组数据,从转录因子结合基序可及性和增强子调控子活性两个层面,揭示了驱动不同DLBCL遗传亚型及其特征生物学模块的转录调控网络。
首先,通过分析肿瘤细胞中转录因子结合基序的可及性,发现其富集模式与特定的遗传亚型和功能模块显著相关。例如,调控浆细胞分化的转录因子基序与ABC相关亚型及浆细胞、增殖模块相关联;而生发中心B细胞相关转录因子基序则与EZB亚型及生发中心模块相关。
更为重要的是,基于SCENIC+算法推断的增强子调控子活性分析,成功地将恶性B细胞亚克隆区分为ABC与GCB两大谱系,并进一步区分了具体的遗传亚型。研究鉴定出大量在特定遗传亚型中活性显著增强或减弱的调控子,它们与对应的B细胞分化模块相关联,并部分由其所在亚型中高频突变的基因编码。
综合分析提出了一个三层级分化轴调控模型:DLBCL遗传亚型的转录状态受调控正常B细胞分化的转录因子驱动,主要沿生发中心B细胞、记忆B细胞和浆细胞这三个主要分化轴向变化。例如,EZB和ST2亚型利用生发中心调控网络;MCD、A53和BN2亚型利用浆细胞调控网络;而N1亚型则利用记忆B细胞调控网络。
此外,研究为关键抑癌基因(如N1和MCD亚型中高频失活的TBL1XR1和ETV6)的功能提供了新见解:它们的抑制性调控子在对应亚型中活性降低,表明其失活促进了这些亚型的恶性表型。
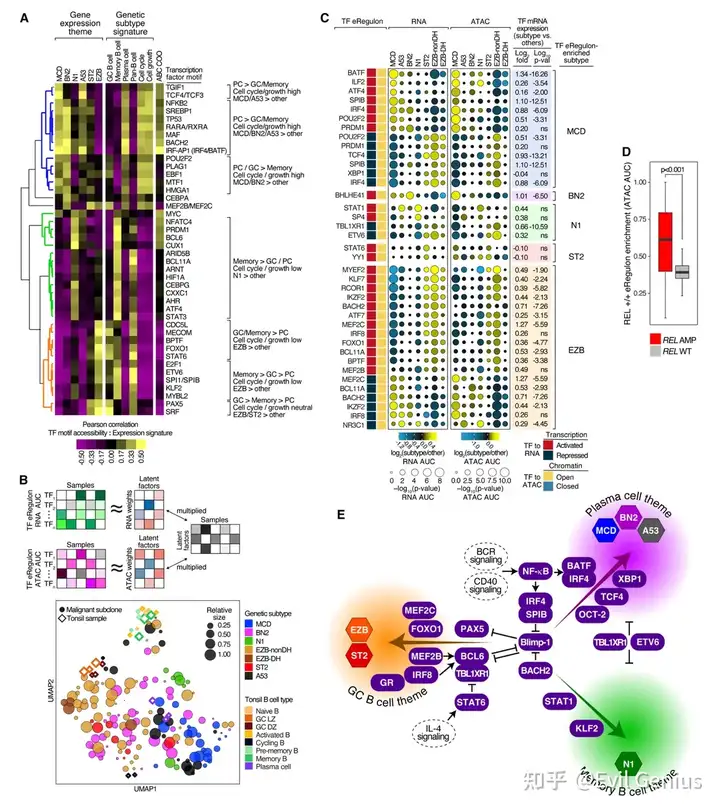
=
看看CNV分析部分


生活很好,有你更好
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
评论
登录后参与评论
推荐阅读
目录

