知识扩展--分子亚型和细胞亚型的异同与联系
原创知识扩展--分子亚型和细胞亚型的异同与联系
原创
追风少年i
修改于 2026-01-29 10:29:12
修改于 2026-01-29 10:29:12
作者,Evil Genius
有人问我持续学习还能坚持多久,我自己也不知道,干一天是一天吧,不用羡慕我这样的人,因为工作生活的不如意才会这样,要是有个安稳的工作,完成工作就好好休息才是人生赢家。
今天我们复习一个内容,首先大家考虑一个问题,同一个组织,做了单细胞 + Xenium/CosMx空间数据,比如CAF细胞,单细胞数据可以依据基因表达进行亚型分析,而Xenium/CosMx空间数据可以依据CAF细胞周围细胞微环境的组成差异进行亚型分析,那么实际中我们应该采用什么策略和方法进行两种亚型的分析整合呢?
如果只有单细胞数据,依据基因表达分亚群即可,只有Xenium/CosMx数据,依据微环境差异分亚型也行,如果我想发一篇好的文章, 同时拥有这两个组学的数据,想多组学联合该怎么做呢?
是单细胞直接分好亚型,transfer到空间上分析其邻域细胞类型的组成特点,还是要Xenium/CosMx依据微环境细分亚型,然后和单细胞的数据对应呢?
多组学难就难在组学之间的联合。
我们先来看看两个组学分亚型的异同。
维度 | 单细胞数据再分群得到的分子亚型 | 空间数据依据“邻域”区分的空间亚型 |
|---|---|---|
定义依据 | 细胞内在的转录组状态。基于所有基因(或高变基因)的表达差异,可能反映:• 不同的功能状态(如激活态、静息态、耗竭态)• 不同的分化阶段或轨迹• 响应不同信号的亚群• 罕见的特殊细胞亚群 | 细胞外在的空间微环境。基于细胞的“邻居类型”和空间位置关系,反映:• 细胞在组织结构中的特定位置(如靠近血管、肿瘤边界、生发中心)• 由特定细胞组合构成的“生态位”或“邻域”• 局部细胞间通信网络的差异 |
数据基础 | 单细胞转录组(scRNA-seq),仅包含基因表达矩阵,丢失空间信息。 | 空间转录组(Xenium, Visium, MERFISH等),包含基因表达和空间坐标。 |
分析方法 | 无监督聚类(如Leiden, Louvain)、轨迹推断、差异表达分析。 | 空间邻域分析(如SpatialDM, Giotto)、细胞邻域富集分析、空间相互作用建模(如CellChat)、空间聚类。 |
揭示的生物学 | 细胞“是什么”和“在做什么”(内在身份与状态)。例(T细胞):CD8+效应T细胞、记忆T细胞、耗竭T细胞、调节性T细胞。 | 细胞“在哪里”和“与谁为邻”(生态位与局部环境)。例(T细胞):肿瘤核心区的T细胞、侵袭前沿的T细胞、三级淋巴结构内的T细胞、基质富集区的T细胞。 |
潜在重叠性 | 同一分子亚型可能分布在多个不同的空间位置。 | 同一空间邻域可能包含来自不同分子亚型的细胞(如果它们被招募或适应同一环境)。 |
核心联系与区别:
联系:空间微环境会强烈影响细胞的转录组状态(例如,靠近血管的癌细胞可能缺氧反应更强)。因此,某些空间亚型可能是由特定的分子亚型主导的。
区别:分子亚型更关注细胞本身固有的特性,而空间亚型更关注由位置决定的外在关系。一个分子亚型可以出现在多个空间生态位(多功能性),而一个空间生态位也可能容纳多个分子亚型(异质性)。
如何对应与整合两种亚型?
将两者对应起来是空间生物学的重要目标,通常遵循以下流程:
步骤1:基础映射与注释
细胞类型注释:首先,对两种数据(单细胞和空间)使用一致的标记基因进行基础的细胞类型注释(如T细胞、巨噬细胞、癌细胞等)。这是后续所有分析的基础。
空间映射:将单细胞数据作为参考,对空间数据的每个细胞/点位进行去卷积或转移标签,获得其基础的细胞类型和可能的分子亚型信息。
步骤2:识别并关联两种亚型
在单细胞数据中定义分子亚型:在已注释的T细胞中,通过聚类和差异表达,识别出T_exhausted, T_effector, T_memory等亚型。
在空间数据中定义空间邻域/生态位:
方法A:细胞邻域分析:对于每个细胞,统计其周围(例如,半径为50μm)内各类邻居细胞的组成比例。然后根据这些“邻居组成特征向量”对所有细胞进行聚类,得到基于微环境的“空间亚型”。
方法B:空间区域分割:使用基因表达和空间信息进行聚类(如BayesSpace),或将组织划分为功能区域(如肿瘤区、基质区、免疫区)。区域内的特定细胞类型即为一种空间亚型。
建立关联:
交叉表分析:创建一个列联表,行是分子亚型,列是空间亚型。检查哪些分子亚型显著富集于哪些空间亚型中。例如,可能发现T_exhausted细胞高度富集在“肿瘤核心-免疫抑制生态位”中。
可视化叠加:在空间图上,用颜色同时标注细胞的分子亚型(点颜色)和其所属的空间生态位(背景色或圈层),直观观察分布规律。
统计检验:使用卡方检验或富集分析(如超几何检验)来量化关联的显著性。
步骤3:机制性整合分析
差异表达分析:在同一个基础细胞类型(如巨噬细胞)内,比较位于不同空间生态位的细胞之间的基因表达差异。这些差异基因可能直接响应微环境信号,是连接空间亚型与分子亚型的关键分子。
例如:比较“血管周围巨噬细胞” vs. “肿瘤坏死区巨噬细胞”的差异基因,可能发现缺氧、血管生成相关通路的激活。
细胞通信分析:针对不同的空间生态位,使用空间分辨的细胞通信工具(如CellChat with spatial info, SpatialDM, MISTy)进行分析。推断驱动特定分子亚型在特定位置聚集或分化的潜在信号通路。
例如:发现“侵袭前沿生态位”中,癌细胞高表达COL1A1,而其周围的成纤维细胞高表达DDR1受体,这种空间特异的配体-受体对可能塑造了该区域癌细胞的独特分子亚型。
多模态整合建模:使用更高级的统计或机器学习模型(如图神经网络、多任务学习),将细胞的转录组特征和其空间邻域特征作为联合输入,预测细胞的状态或功能,从而得到一个统一的分型框架。
分子亚型 | 空间亚型 | 整合视角 | |
|---|---|---|---|
核心 | 细胞内在状态 | 细胞外在位置与邻居 | 细胞是内在状态与外在环境相互作用的动态产物 |
对应策略 | 作为“种子”映射到空间 | 作为“容器”分析其内容物 | 构建“分子亚型 ↔ 空间生态位”的富集网络 |
终极目标 | 理解细胞的功能特性 | 理解组织的结构功能单元 | 揭示驱动特定细胞在特定位置呈现特定状态的时空调控程序,为精准治疗提供靶点。 |
基于以上认识,我们来看看实际的例子,王凌华教授给我们的答案。
文章是

首先来看细胞亚型,也就是空间亚型,依据空间微环境不同分析好亚型找基因表达差异。
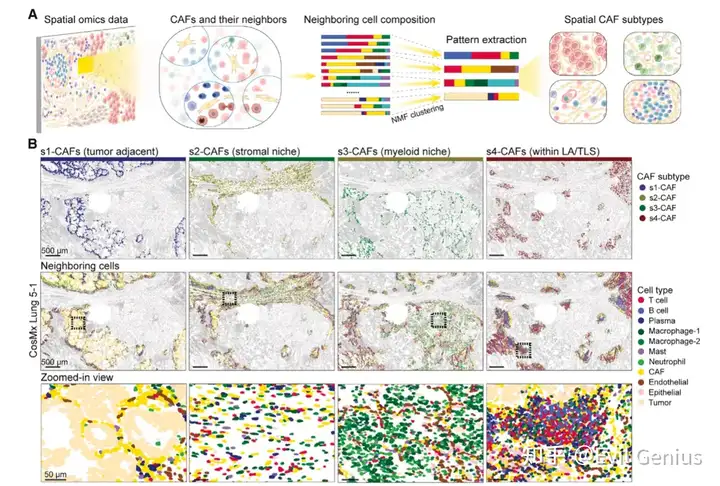
分析方法

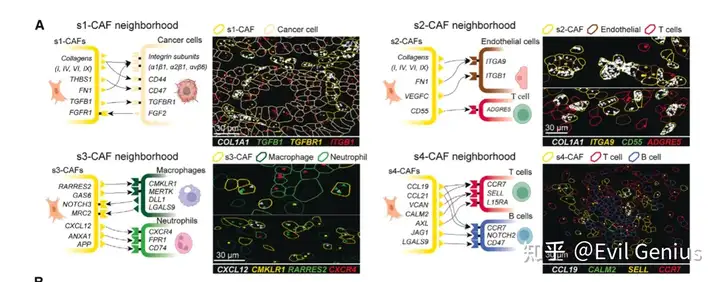
再来看看分子亚型,也就是单细胞数据分亚型,然后分析空间微环境差异。
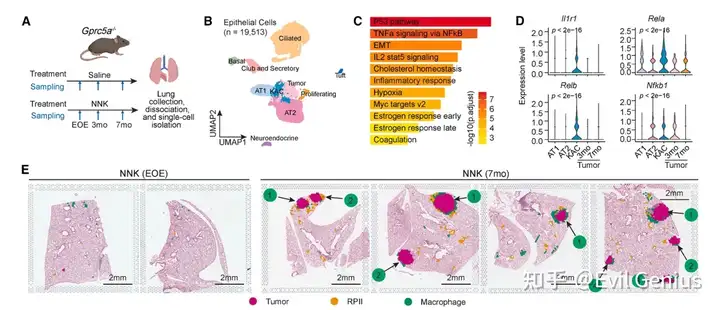
看看方法


那么有没有可能匹配的单细胞数据分好的亚型与空间微环境分好的亚型能够直接对应上呢?
我也期待这样的文章给我们启发。
生活很好,有你更好。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
评论
登录后参与评论
推荐阅读
目录

