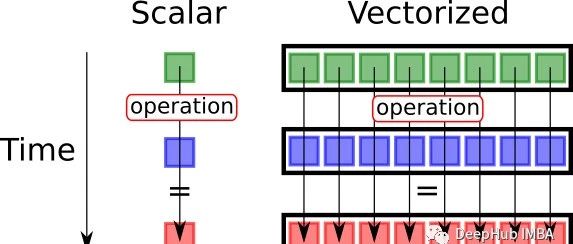
多元正态分布的Numpy向量化
多元正态分布的Numpy向量化
提问于 2018-02-08 13:42:38
我有两个二维numpy数组A,B。我想使用scipy.stats.multivariate_normal来计算A中每行的联合logpdf,使用B中的每一行作为协方差矩阵。有没有什么方法可以在不显式循环行的情况下做到这一点?将scipy.stats.multivariate_normal直接应用于A和B确实会计算A中每一行的logpdf (这是我想要的),但使用整个2D数组A作为协方差矩阵,这不是我想要的(我需要B的每一行创建一个不同的协方差矩阵)。我正在寻找一种使用numpy向量化并避免在两个数组上显式循环的解决方案。
回答 1
Stack Overflow用户
发布于 2018-06-12 03:57:39
我也在尝试完成类似的事情。下面是我的代码,它接受三个NxD矩阵。X的每一行是一个数据点,means的每一行是一个均值向量,covariances的每一行是一个对角协方差矩阵的对角向量。结果是对数概率的长度为N的向量。
def vectorized_gaussian_logpdf(X, means, covariances):
"""
Compute log N(x_i; mu_i, sigma_i) for each x_i, mu_i, sigma_i
Args:
X : shape (n, d)
Data points
means : shape (n, d)
Mean vectors
covariances : shape (n, d)
Diagonal covariance matrices
Returns:
logpdfs : shape (n,)
Log probabilities
"""
_, d = X.shape
constant = d * np.log(2 * np.pi)
log_determinants = np.log(np.prod(covariances, axis=1))
deviations = X - means
inverses = 1 / covariances
return -0.5 * (constant + log_determinants +
np.sum(deviations * inverses * deviations, axis=1))请注意,此代码仅适用于对角线协方差矩阵。在这种特殊情况下,下面的数学定义被简化了:行列式变成元素的乘积,逆变成元素的倒数,矩阵乘法变成元素的乘法。

快速测试正确性和运行时间:
def test_vectorized_gaussian_logpdf():
n = 128**2
d = 64
means = np.random.uniform(-1, 1, (n, d))
covariances = np.random.uniform(0, 2, (n, d))
X = np.random.uniform(-1, 1, (n, d))
refs = []
ref_start = time.time()
for x, mean, covariance in zip(X, means, covariances):
refs.append(scipy.stats.multivariate_normal.logpdf(x, mean, covariance))
ref_time = time.time() - ref_start
fast_start = time.time()
results = vectorized_gaussian_logpdf(X, means, covariances)
fast_time = time.time() - fast_start
print("Reference time:", ref_time)
print("Vectorized time:", fast_time)
print("Speedup:", ref_time / fast_time)
assert np.allclose(results, refs)我得到了250倍的加速。(是的,我的应用程序要求我计算16384种不同的高斯分布。)
页面原文内容由Stack Overflow提供。腾讯云小微IT领域专用引擎提供翻译支持
原文链接:
https://stackoverflow.com/questions/48686934
复制相关文章
相似问题
Numpy:多元正态分布的对数可能性
多元numpy不是正态分布,请帮助纠正这个错误
随机多元正态分布
聚类多元正态分布
是否有一种从Numpy的多元正态分布中以矢量化方式进行批量抽样的方法?
社区富文本编辑器全新改版!诚邀体验~
全新交互,全新视觉,新增快捷键、悬浮工具栏、高亮块等功能并同时优化现有功能,全面提升创作效率和体验